La prima mappa del genoma umano, 20 anni fa
Il 14 aprile del 2003 il Progetto genoma umano annunciò di avere sequenziato buona parte del DNA umano, uno dei più grandi progressi nella storia della biologia

Il 14 aprile di venti anni fa il Progetto genoma umano (Human Genome Project, HGP) annunciò di avere completato il primo sequenziamento del genoma umano. L’annuncio arrivava dopo più di dieci anni di ricerche nell’ambito di una delle più grandi collaborazioni internazionali mai realizzate nel campo della biologia. Da allora i gruppi di ricerca hanno scoperto molte altre cose sul DNA e soprattutto sono riusciti a sequenziare le parti che per motivi pratici e tecnici erano state ignorate nel grande lavoro di mappatura svolto dal Progetto genoma umano. Quella grande attività di ricerca fu comunque essenziale per scoprire meglio perché siamo fatti in un certo modo e talvolta ci ammaliamo.
Il genoma è tutto il DNA che troviamo all’interno di una cellula. Possiamo considerarlo come il manuale di istruzioni di una cellula: contiene tutte le informazioni per produrre le proteine che costituiscono il nostro organismo e che lo fanno funzionare.
Premessa: cos’è il DNA
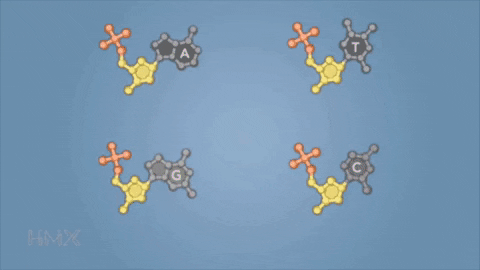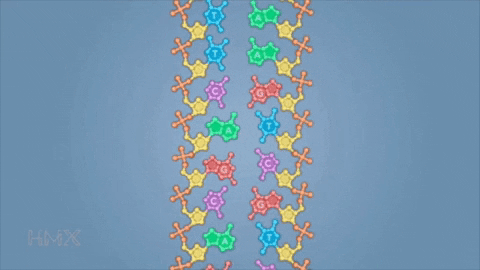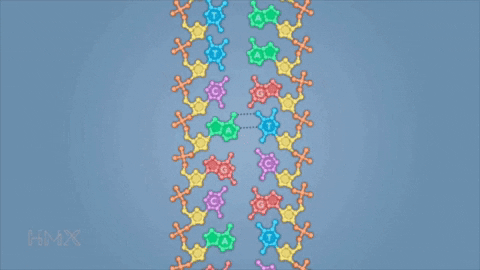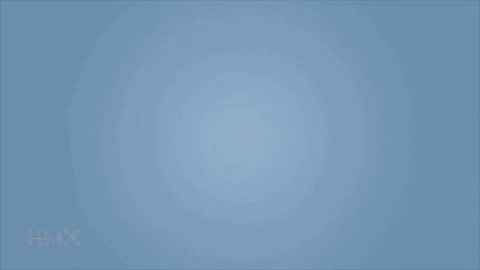
Il DNA è costituito dai nucleotidi, che a loro volta sono composti da zucchero, gruppi fosfato e una di queste quattro basi azotate: adenina (A), guanina (G), citosina (C) e timina (T). Zucchero e gruppi fosfati sono concatenati tra loro e costituiscono l’impalcatura di un lungo filamento del DNA; le basi azotate si uniscono tra loro portando alla formazione del doppio filamento. A si lega con T, mentre C si lega con G. La sequenza di queste lettere determina l’informazione genetica, ciò che serve in sostanza per determinare forma e funzione delle cellule.
(Harvard University)
Le cellule possono essere molto diverse tra loro, ma nel loro nucleo contengono tutte il medesimo genoma, costituito da 3 miliardi di coppie di basi azotate (altro DNA è contenuto nei mitocondri ed era già stato mappato in precedenza). Se potessimo stendere tutto il DNA contenuto in una cellula otterremmo un filo lungo quasi due metri. Nel nucleo della cellula questo materiale genetico è organizzato per occupare poco spazio, attorcigliato su se stesso e condensato in una fitta e microscopica matassa.
Il DNA nucleare si organizza in 23 coppie di cromosomi, che possiamo immaginare come matasse più piccole a forma di X (fatta eccezione del cromosoma Y nei maschi) per la trasmissione delle informazioni genetiche alle nuove cellule. Le istruzioni vere e proprie, cioè i geni, rappresentano circa il 2 per cento del genoma: 20mila geni coinvolti nella produzione di 100mila tipi diversi di proteine.
Il restante 98 per cento era considerato fino a poco tempo fa inutile, tanto da essere definito junk DNA, cioè DNA spazzatura. Si pensava che non servisse praticamente a nulla e che fosse ciò che rimaneva dei processi evolutivi che ci hanno portato fino ai giorni nostri. Negli ultimi anni si è invece scoperto che quel materiale non è spazzatura e che anzi ha un ruolo importante nella regolazione dell’espressione dei geni, nel tenere in ordine il DNA stesso e in altre attività.

Cromosomi umani (Wikimedia)
L’idea di sequenziare il DNA
L’esistenza del genoma non era certo un mistero, ma per lungo tempo la sola idea di mappare tutti quei miliardi di sequenze di A, T, C e G sembrava uno sforzo improbabile, se non irrealizzabile. C’era però la consapevolezza dell’importanza di determinare la sequenza di quelle coppie di basi azotate, in modo da identificare e mappare i geni per studiare meglio il nostro organismo, le sue funzioni e soprattutto le anomalie nel materiale genetico (o nel suo impiego come manuale di istruzioni) che possono portare a malattie di vario tipo e difficili da curare.
A metà degli anni Novanta alcuni importanti ricercatori iniziarono a ipotizzare l’organizzazione di ambiziosi programmi di ricerca per provare a sequenziare tutto il genoma umano. Tra loro c’era anche il biologo italiano Renato Dulbecco, premio Nobel per la medicina nel 1975, che aveva proposto l’idea dell’intero sequenziamento in un saggio pubblicato su Science, una delle più importanti riviste scientifiche al mondo. La sua idea e altre proposte simili presentate in ricerche scientifiche e convegni portarono infine all’istituzione del Progetto genoma umano, uno dei più grandi mai realizzati nel campo della biologia con un finanziamento stimato intorno ai 3 miliardi di dollari.
Il Progetto genoma umano, o HGP
Il progetto fu lanciato ufficialmente nel 1990 con lo scopo di sequenziare tutto il DNA contenuto nelle cellule di un essere umano. Nell’iniziativa furono coinvolti oltre 20 centri di ricerca in giro per il mondo, con una maggiore concentrazione negli Stati Uniti dove erano state impegnate più risorse. I campioni furono prelevati da una ventina di volontari, in modo da avere una mappatura un minimo rappresentativa di un essere umano medio. Il DNA umano è al 99,9 per cento identico in tutti gli individui, ma ciò che resta è più che sufficiente – insieme ad altri fattori come quelli ambientali – per far sì che ciascuno di noi sia diverso.
Le attività di ricerca procedettero velocemente, anche perché il progetto pubblico doveva fare i conti con la concorrenza della società statunitense Celera Genomics, che aveva annunciato di voler sequenziare l’intero genoma umano in pochi anni. Nel 2000 HGP pubblicò una prima versione ancora piuttosto incompleta del genoma, occasione che fu sfruttata dall’allora presidente degli Stati Uniti, Bill Clinton, per comunicare l’importante risultato di persona.
In una conferenza stampa cui partecipò anche l’allora primo ministro britannico Tony Blair, Clinton utilizzò toni trionfalistici dicendo che: «Grazie a questa nuova profonda conoscenza, il genere umano è a un passo dal guadagnare un nuovo immenso potere per la guarigione. La scienza genomica avrà un impatto reale su tutte le nostre vite e ancora di più su quelle dei nostri bambini. Rivoluzionerà la diagnosi, la prevenzione e il trattamento di molte, se non tutte, le malattie che interessano gli esseri umani».
Tre anni dopo, i responsabili del Progetto genoma umano dichiararono completo il lavoro di sequenziamento, avendo raggiunto la mappatura del 90 per cento del genoma: con le tecnologie disponibili all’epoca, era stato pressoché raggiunto il limite di ciò che si potesse analizzare.
Il DNA ripetuto
I gruppi di ricerca di HGP non erano infatti riusciti a mappare le sequenze ripetute di DNA. Alcune parti del DNA ripetono la stessa sequenza di basi anche migliaia di volte. Le ripetizioni sono presenti in particolar modo sui telomeri, cioè le quattro estremità di ogni cromosoma (che come abbiamo visto hanno una forma che ricorda una X), e nei centromeri, la parte centrale del cromosoma.
Non è ancora molto chiaro quale sia la funzione delle numerose ripetizioni, ma il fatto che si trovino in quei punti suggerisce che servano a proteggere il resto del DNA quando avviene la divisione cellulare, evitando che se ne perdano dei pezzi importanti con le relative informazioni. A ogni replicazione i telomeri si accorciano e si ipotizza che questo processo sia associato all’invecchiamento dei tessuti, con la loro progressiva perdita di funzionalità. I centromeri sono altrettanto importanti nella divisione cellulare ed è stato osservato che il loro malfunzionamento è coinvolto nello sviluppo di alcune forme di tumore.
Il metodo impiegato dal Progetto genoma umano, con i mezzi disponibili all’epoca, non consentiva di sequenziare il DNA ripetuto. Il sistema non prevedeva infatti di leggere tutto il genoma dall’inizio alla fine, ma di spezzarlo in pezzi più piccoli contenenti ciascuno alcune centinaia di coppie di basi. Per ogni pezzo veniva letta la sequenza di A, T, C e G presente e si procedeva poi a rimettere i pezzi insieme attraverso un programma al computer. Era un approccio piuttosto efficiente, ma che non poteva funzionare con le sequenze ripetute, perché non c’era modo di capire in che ordine dovessero essere rimessi insieme i pezzi che le contenevano, visto che erano sempre le stesse lettere a ripetizione.
Dopo il Progetto genoma umano
Alla versione ufficiale presentata venti anni fa mancava quindi un pezzo, circa l’8 per cento del genoma umano: il manuale delle istruzioni era quasi completo, ma rimanevano oltre 400 interruzioni che avrebbero richiesto circa vent’anni per essere colmate. Grazie alla disponibilità di alcune nuove tecnologie e al lavoro di gruppi di ricerca più piccoli, non coinvolti in un progetto dalle grandi dimensioni come quello avviato negli anni Novanta, lo scorso anno è stato annunciato un importante progresso nella mappatura pressoché integrale del genoma. Il lavoro è ancora in corso, ma potrebbe essere concluso entro quest’anno riempiendo le lacune che il progetto originario non aveva potuto affrontare.
Lo studio delle sequenze di DNA che mancavano e di quelle mappate dal Progetto genoma umano ha svelato una complessità ancora più grande di quanto si aspettassero i gruppi di ricerca, portando a più domande che risposte. L’analisi ha permesso di identificare lo scopo di alcuni geni, ma al tempo stesso ha reso più evidente come determinate forme e funzioni nel nostro organismo derivino dal concorso di più geni, dai fattori ambientali, dalle abitudini che ciascuno di noi ha e molto spesso da circostanze del tutto casuali e difficili da sistematizzare.
Quando Clinton fece le sue dichiarazioni entusiastiche, si pensava ancora che per eliminare alcune malattie fosse sufficiente “spegnere” un gene, mentre oggi sappiamo proprio grazie agli studi sul genoma che è tutto molto più complicato. Maggiori complicazioni non implicano comunque l’impossibilità di sviluppare terapie geniche personalizzate, un ambito della ricerca in piena espansione e che sta portando alle prime applicazioni pratiche, per ora molto costose, ma promettenti. Ulteriori progressi sono stati resi possibili dalle tecniche di modifica del DNA, come CRISPR/Cas-9 che ha rivoluzionato interi settori della ricerca, aprendo numerose questioni etiche in parte ancora da risolvere.
– Ascolta anche: La puntata di “Ci vuole una scienza” sulle terapie geniche
Negli anni in cui era in corso, il Progetto genoma umano fu molto discusso e commentato non solo in ambito scientifico, ma anche politico e di regolamentazione sui dati che avrebbe prodotto. Con non poche difficoltà, anche a causa della concorrenza da parte di un’azienda privata come Celera Genomics, si decise infine che le sequenze di DNA dovessero essere di pubblico dominio e consultabili liberamente da tutti. Il programma di ricerca ebbe il merito di promuovere un approccio più aperto su alcuni ambiti della genetica, evitando che certe informazioni rimanessero a disposizione di pochi con il rischio di ridurre le opportunità di studio e il libero accesso a dati molto importanti per i gruppi di ricerca.
HGP contribuì inoltre a rendere più presenti la genetica e le sue applicazioni nel dibattito pubblico, seppure con qualche fraintendimento sul genoma e sulla sua completa mappatura. Come abbiamo visto non esiste un genoma soltanto: il materiale genetico di ogni individuo è diverso e quelle differenze, per quanto limitate, contano molto nel renderci unici. Per questo i responsabili del progetto originario provarono a mappare un genoma “medio”, utilizzando campioni provenienti da vari volontari. I partecipanti erano per lo più della zona di New York, un’area geograficamente limitata per fornire un genoma rappresentativo, ammesso che se ne possa ottenere uno con questa caratteristica.
Per provare a superare queste limitazioni, da qualche anno è stato avviato lo Human Pangenome Reference Consortium, un consorzio che ha l’obiettivo di sequenziare il DNA di centinaia di persone da posti molto diversi del pianeta. Il lavoro di mappatura ha permesso di descrivere già 47 genomi che sono stati poi combinati insieme per realizzare una prima versione di un “pangenoma”. Il progetto è solo all’inizio e potrebbe diventare una costante della ricerca, considerato che il genoma stesso non è statico, ma cambia nel corso del tempo man mano che passano le generazioni.
Avere un genoma “medio” di riferimento è comunque importante per poter fare i confronti con quelli dei singoli individui. Grazie ai progressi nei sistemi automatici per il sequenziamento, diventerà sempre più semplice ed economico fare la mappatura di un genoma, potenzialmente uno dei primi passi per terapie geniche personalizzate.





